
Idee
Mithilfe des K-Means clustering Algorithmus lernt der Computer, Blätter verschiedener Baumarten zunächst zu unterscheiden und danach auch zu bestimmen.
Dieses Projekt bietet vielfältige Gelegenheiten, Neues zu lernen und sich sowohl in der Programmierung als auch der Projektorganisation zu üben.
Funktion
Das Projekt implementiert und demonstriert die Funktionsweise des K-Means-Algorithmus zur Gruppierung (clustering) von Datensätzen. Dieser Algorithmus bietet im Bereich der Datenanalyse vielfache Anwendungsmöglichkeiten und hat daher eine weite Verbreitung gefunden.
Für dieses Projekt verwenden wir Datensätze aus einer anschaulichen Quelle, nämlich den Fotografien von Pflanzenblättern. Diese könnten wir aus heimischen Wäldern selber erstellen oder aber aus einer schon vorhandenen Datenbank schöpfen. Wir verwenden die Folio-Daten. Natürlich lässt sich die Implementierung des Algorithmus mit beliebigen anderen Datensätzen nutzen.
Wir verfahren wie folgt:
- Wir wählen von 7 Pflanzenarten (siehe Bild oben) jeweils 15 Exemplare d.h. Fotos von Blättern.
- Aus jedem Foto extrahieren wir 5 Kennzahlen (features), welche die Form des Blattes beschreiben.
- Diese 5 Zahlen können wir uns als Koordinaten eines Vektors in einem 5-dimensionalen Hyperraum vorstellen.
- Die Vektoren der Blätter jeder Pflanzenart (Spezies) liegen nahe beieinander und getrennt von den Vektoren anderer Arten. Sie bilden sogenannte Cluster (Haufen).
- Das K-Means-Verfahren findet diese Cluster und gibt uns deren geometrischen Mittelpunkt (Centroid).
- Das Clustering können wir als Lernprozess begreifen und ihn dazu benutzen, weitere Blätter zu klassifizieren. Dazu messen wir den Abstand ihres Kennzahlen-Vektors zu den Centroiden. Der nächstgelegene Centroid bestimmt dann die Spezies des vorgelegten Blattes.
Wir verwenden keine vorgefertigten Python-Pakete zum maschinellen Lernen, sondern nur Python Standard-Pakete wie Math, NumPy und Matplotlib, sowie PIL zur Bild-Aufbereitung. Die Visualisierung spielt eine große Rolle für das Verständnis des Algorithmus, sowohl was seine Funktion als auch seine Grenzen angeht. Die folgenden Bilder zeigen dies.
Nicht alle Blätter eine Baumart sehen gleich aus, wie diese fünf Exemplare von Chrysanthemum belegen:

Aus den Fotos gewinnen wir Schwarz-weiß-Bilder: Jeder Bildpunkt, der zum Blatt gehört, ist schwarz, alle anderen sind weiß:

Um die Zerklüftung der Blattkontur zu messen, müssen wir den Umriss erst einmal ermitteln. Wir programmieren dazu den Marching-Squares-Algorithmus:
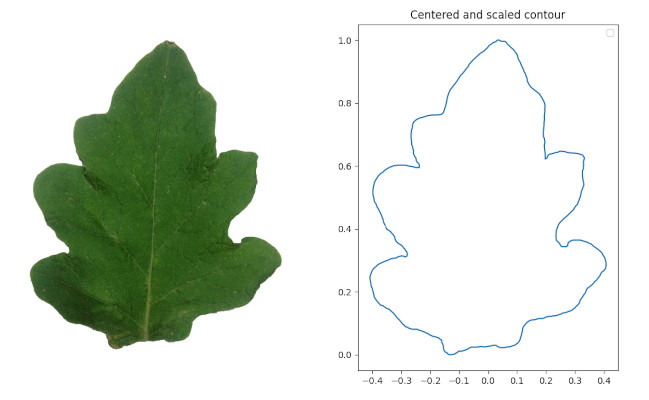
Danach ermitteln wir 5 Kennzahlen zu jedem Blatt (die genaue Beschreibung findet sich in der Dokumentation) und können uns die so entstehenden Punktwolken anzeigen lassen. Achtung: Hier werden nur zwei der fünf Features verwendet („belly“ und „fill“).
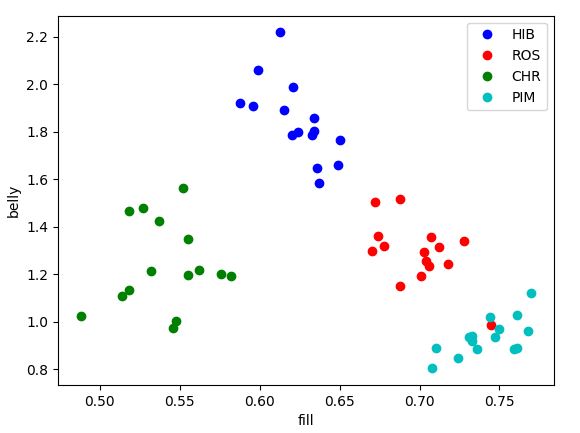
Trotz der Unterschiede der einzelnen Exemplare einer Spezies untereinander erkennt man doch, dass die abgeleiteten Features eine Zusammengehörigkeit erkennen lassen. Wie man an dem roten „Ausreißer“ sieht, sind die Haufen aber nicht immer klar begrenzt. Die anderen 3 Spezies sind hier nicht dargestellt, weil sie sich mit den dargestellten überschneiden.
Bei einem Clustering-Lauf geschieht folgendes: Dem K-Means-Algorithmus werden 105 Datensätze zu je 5 Zahlenwerten in zufälliger Reihenfolge vorgelegt. Es gelingt ihm, die 7 zunächst zufällig gewählten Centroiden im 5-dimensionalen Hyperraum so zu verschieben, dass sie im geometrischen Mittelpunkt der artspezifischen Cluster landen. Das ist erstaunlich, zumal wir uns einen 5-dimensionalen Raum gar nicht vorstellen können.
Die Bahnen der Centroiden während der Ausführung des K-Means-Algorithmus zeigt das folgende Bild: Gegeben seien drei Punktwolken im 3-D-Raum: Rot, Blau und Grün, die wir uns in einem Quader platziert vorstellen. Mit dem interaktiven 3D-Scatter-Plot, wie Matplotlib ihn anbietet, kann man die Situation gut erkunden:
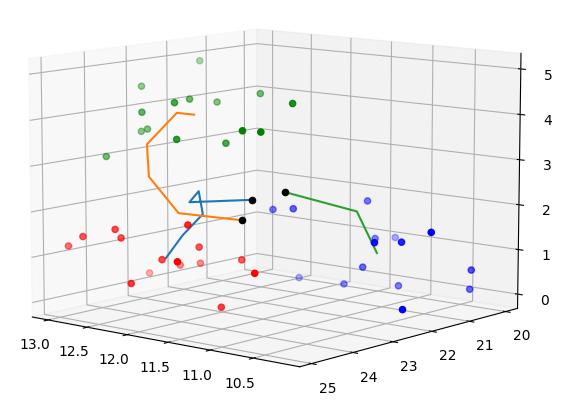
Obwohl K-Means die Farben ja nicht sieht, finden die Centrioden ihren Weg in die Cluster. Beginnend mit Startwerten etwa in Bildmitte (durch schwarze Punkte markiert) wandert Centroid 1 (blaue Spur) ins Rot, Centroid 3 (grüne Spur) geht ins Blau, während Centroid 2 (orange Spur) zum Grün hinaufsteigt. Den räumlichen Eindruck erhält man mit dem „lebendigen“ Plot, wenn man sich mit der Maus verschiedene Perspektiven auf den Quader verschafft, was hier auf der Website leider nicht geht.
Doch zurück zu unserer Aufgabe: Nach einem erfolgreichen Clustering-Lauf werden die Blätter einer Spezies also jeweils einem Centroiden zugeordnet. Dies ist die textuelle Ausgabe des Programms cluster.py:
Summary table:
Cluster 1 : JAC(15) PIM(2)
Cluster 2 : ROS(15)
Cluster 3 : EGG(15)
Cluster 4 : PIM(13) FRU(1)
Cluster 5 : HIB(15)
Cluster 6 : FRU(14)
Cluster 7 : CHR(15)
3 leaves out of 105 were wrongly categorized
Performance ratio = 97.1 %
Wie man erkennt, finden die 7 Centroiden zu den vorhandenen Häufungen d.h. Clusters der Spezies. Allerdings werden 3 Blätter nicht den korrekten Centroiden zugeordnet. Dies liegt daran, dass die 5 verwendeten geometrischen Features nicht zur eindeutigen Erkennung ausreichen.
Eine Besonderheit des K-Means-Algorithmus liegt darin, dass man die Anzahl der Cluster (Parameter k) vorgeben muss. Falls k unbekannt ist, führt man den Algorithmus mit verschiedenen Werten für k aus und berechnet nach jedem Durchlauf des Algorithmus den mittleren Abstand der Blätter von den jeweiligen Centroiden. Dann kann man sich leicht vorstellen, dass für k=1 dieser Abstand maximal ist. Versteht man als anderes Extrem jeden einzelnen Datenpunkt als Cluster, wird der mittlere Abstand zu Null. Damit erzielen wir jedoch keinerlei Erkenntnisgewinn. Irgendwo zwischen diesen Extremen wird also das optimale k liegen müssen.
Trägt man den mittleren Abstand als Funktion von k auf, erhält man das sogenannte Ellenbogen-Diagramm. Es macht einen Knick beim optimalen k, hier bei k=7:
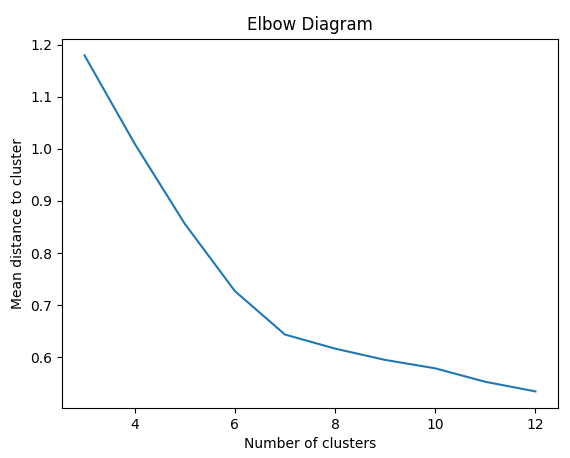
Aus diesem Diagramm lässt sich leicht das optimale k als Maximum der Steigungsdifferenzen aufeinanderfolgender k-Intervalle ablesen:
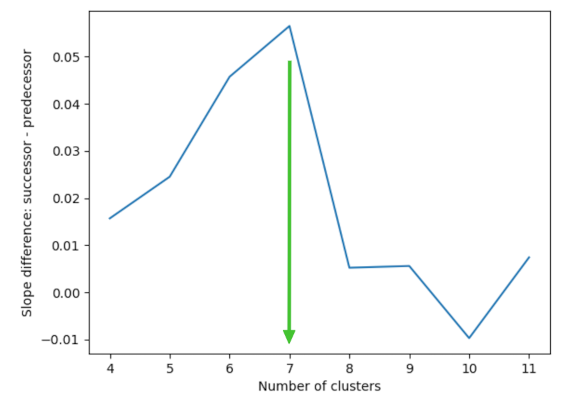
Mit diesem Verfahren ermitteln wir die 7 als korrekte Anzahl der Spezies. (Beachte, dass damit die Spezies natürlich nicht benannt sind!)
Begreift man den Clustering-Prozess als maschinelles Lernen, dann sollte es auch dazu in der Lage sein, bislang nicht berücksichtigte Bilder von Blättern richtig einzuordnen. Dies wurde mit je 3 Blättern pro Spezies getestet mit dem Resultat, dass von 21 Blättern nur eines falsch einsortiert wurde.
Eine tiefer gehende Dokumentation findet sich hier.
Wer selbst programmieren möchte, kann meine Programme zur Anregung nutzen. Die Daten liefere ich hier gleich mit.
